Ledock_omega 通过显性水提高分子对接准确性
从学术免费版开放以来,ledock已被越来越多的学术用户重视,而学术专业版ledock_go对其准确性和速度进行了更大的调整,并增加了sdf格式的支持,新冠疫情期间,为提升其对有核酸参与的靶点的对接,进行了进一步调整,使得其研究对象进行了拓宽。ledock本身采用的是模拟退火和遗传算法进行构象搜索,ledock_go则采用了模拟退火和最陡下降的算法,提升了其速度,也适合在单cpu和单核使用。最近ledock进一步改进提出了ledock_omega版本,在具有显水信息的帮助下进一步提升其对接准确性,建议对结构和药物设计有一定经验的用户使用。
ledock_omega版本需要水分子的信息,因此第一步是预测水分子的位置,以及替代水分子的能量,氢键贡献等信息,这里采用的是lesite程序,受体文件的输入格式仍然为charmm27兼容的pdb文件,可以采用lepro生成,也可以采用其他程序生成,只要原子类型兼容该版本的力场即可。需要注意的是,无论是ledock和lesite,其氢键的计算对氢原子的构象比较敏感,建议在使用之前对H原子进行优化,甚至有些情况需要对His,Gln,Asn的侧链进行flip操作。这里介绍一般的操作流程,但并不代表以上的建议不需要。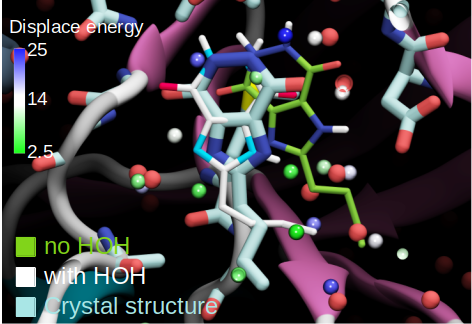
1.下载pdb
wget https://files.rcsb.org/download/1n2v.pdb2.采用lepro生成charmm27兼容的pro.pdb文件,以及分子对接需要的参数文件dock.in文件
lepro 1n2v.pdb -metal3.采用lesite预测pro.pdb中口袋(根据dock.in中的口袋定义)的水分子的位置和能量信息,生成waters.pdb文件
lesite dock.in4.这里不介绍ledock的具体教程,小分子sdf/mol2文件自行准备,采用ledock_omega进行对接
ledock_omega dock.in -water从上面的结果来看,如果不给出蛋白上结合水的信息,小分子的对接位置偏离晶体结构中的位置,这大概是众多分子对接软件都会有的问题,由于采用显水,并且参考其置换水的能量,可以看出ledock_omega给出的结果与晶体结构中的位置是一致的。水分子在分子识别中的作用在许多研究论文中都有阐述和介绍,然而这一信息并不是那么容易获得,即使是watermap也难免有不理想的情况,对于lesite以及ledock_omega的使用,也要适当地参考水分子,例如对于锚定位点的水分子(金属离子结合的水,关键位点氢键形成的点),这里并不建议保留,仍然需要研究者对于研究对象有较深的理解。但lesite和ledock_omega做到了易用性和准确性的平衡。水能载舟,亦能覆舟。分子对接这一基本的工具如何用好水,这里给出了一个典型的案例。Ledock对于分子对接方面的思想也一直在改进,帮助研究者更好地设计药物!